Artigo científico da UFTM mostra como uma biomolécula poderia inibir a infecção por coronavírus
Um estudo conduzido pela Universidade Federal do Triângulo Mineiro (UFTM), em colaboração com a Universidade Estadual de Campinas (Unicamp), investigou a interação de um peptídeo inibidor do coronavírus, agindo na superfície do vírus, mais precisamente na coroa dele, mais conhecida como proteína spike. Peptídeos são compostos formados pela união de dois ou mais aminoácidos (moléculas orgânicas).
O trabalho teórico demonstrou, por técnicas computacionais, que são completamente diferentes os modos de interação dos peptídeos estudados com o novo coronavírus de 2019 (altamente transmissível e mortal) e com a sua versão original de 2002. O resultado está de acordo com trabalhos experimentais. Essa descoberta demonstra o potencial do protocolo teórico aplicado em triagens de peptídeos mais potentes que poderiam compor novas terapias para controle e vigilância da pandemia da COVID-19, sendo as triagens baseadas em simulações computacionais (virtual screening).
Os resultados foram publicados em um artigo científico na revista Journal of Chemical Information and Modeling, um periódico científico publicado pela American Chemical Society, com foco na química computacional.
A pesquisa
A infecção da doença acontece quando a proteína spike, da superfície do coronavírus (SARS-CoV-1 de 2002 e SARS-CoV-2 de 2019), liga-se à enzima receptora da célula humana, chamada enzima conversora de angiotensina 2 (ECA2). Uma estratégia do estudo para evitar a infecção do vírus seria projetar peptídeos no ambiente celular que se ligariam rapidamente e fortemente à proteína do coronavírus, driblando a ligação dele com a ECA2 humana, evitando a entrada do vírus nas células hospedeiras.
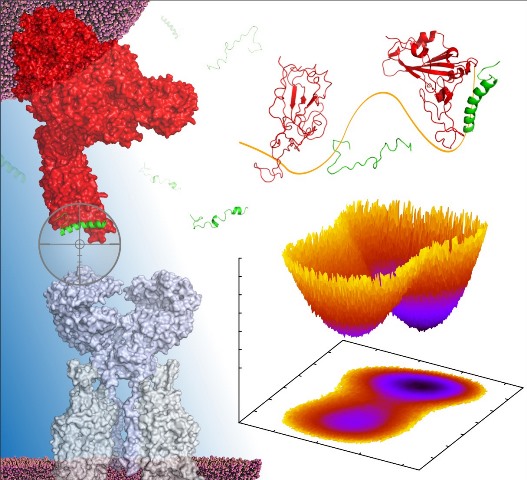
Tendo em vista essa tática, a equipe da pesquisa desenhou no computador um peptídeo híbrido baseado no peptídeo natural da ECA2 e demonstraram os desdobramentos de uma interação com SARS-CoV-1 e SARS-CoV-2, em estudos experimentais. "A energia de interação da spike do coronavírus é mais favorável com o peptídeo híbrido desenhado do que com o peptídeo natural", aponta Paulo Henrique Borges Ferreira, um dos autores da pesquisa, recentemente formado pelo Programa de Pós-Graduação em Química da UFTM.
Por conta disso, os pesquisadores testaram, por simulação computacional, se o peptídeo híbrido desenhado teria também uma afinidade aumentada para SARS-CoV-2. Os resultados não só comprovaram a hipótese levantada como também demonstraram que os peptídeos inibidores teriam um novo mecanismo para bloquear a ação do SARS-CoV-2. Isso sugere uma possível correlação com a forma mais infecciosa do novo coronavírus.
“É muito interessante notar que a nova região de ligação da spike que surgiu como resultado do artigo é a mesma região em que apareceram as novas mutações, ainda mais infecciosas, descobertas recentemente. Isso demonstra a importância de se ter protocolos computacionais que trabalhem em conjunto com grupos experimentais para rapidamente desenvolver biomoléculas contra novas variantes do vírus”, diz Ronaldo Júnio de Oliveira, professor do Departamento de Física da UFTM e coordenador do estudo.
Para os pesquisadores envolvidos, esse estudo teórico será um apoio importante ao estudo clínico, podendo inclusive aprimorar e acelerar etapas em pesquisas de terapias. "O protocolo computacional obteve sucesso em determinar não só a intensidade com a qual os peptídeos testados se ligavam à proteína spike, como também permitiu a análise de alguns mecanismos de ligação. A metodologia pode ser utilizada para um teste rápido e de baixo custo de futuros compostos a serem utilizados para minimizar ou mesmo evitar a infecção pelo SARS-CoV-2 e suas variantes", aponta Frederico Campos Freitas, recentemente formado pelo Programa de Pós-Graduação em Química da UFTM e técnico dos laboratórios de Física.
O estudo foi feito com a colaboração de Denize Cristina Fávaro, professora do Instituto de Química da Unicamp. Os recursos para a pesquisa são provenientes de outros projetos de pesquisa aprovados pelos professores Ronaldo e Denize nas agências financiadoras FAPEMIG, FAPESP, Capes e CNPq, e em editais de fomento à pesquisa das próprias instituições sede UFTM e Unicamp.
O artigo "Shedding Light on the Inhibitory Mechanisms of SARS-CoV-1/CoV-2 Spike Proteins by ACE2-Designed Peptides" pode ser lido em https://doi.org/10.1021/acs.jcim.0c01320.
Mais informações sobre o laboratório de pesquisa podem ser encontradas em https://ronaldolab.github.io.
Imagem do destaque: Estrutura do peptídeo (verde) desenhado no computador inibindo a ação da proteína spike do coronavírus (vermelho).

Redes Sociais