Docente da Física participa de pesquisa sobre técnica de simulação de cromossomos publicada em artigo científico internacional
O periódico científico The Proceedings of the National Academy of Sciences (PNAS), uma das revistas científicas multidisciplinares mais citadas e abrangentes do mundo, publicou no final de janeiro de 2026 o artigo científico A data-driven chromatin model reveals spatial and dynamic features of genome organization (Um modelo de cromatina baseado em dados revela características espaciais e dinâmicas da organização do genoma), sendo um dos autores o professor Ronaldo Oliveira, do Departamento de Física da UFTM.
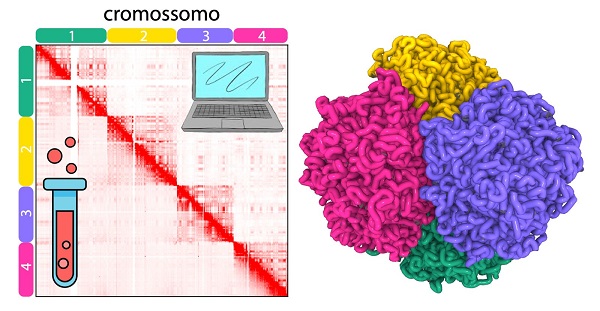
A pesquisa experimentou técnicas de simulação de cromatina, que representam os cromossomos, uma mistura de DNA e proteínas localizadas dentro do núcleo das nossas células. A compactação da cromatina dentro do núcleo celular representa um desafio significativo para a biologia. Como o núcleo celular é extremamente pequeno, a natureza desenvolveu mecanismos altamente eficientes para compactar esse material. A cromatina é, essencialmente, esse DNA todo organizado, enrolado e compactado. Pesquisas sobre o funcionamento desse processo, esse sistema biológico, promovem avanços fundamentais para entender como a forma 3D do genoma controla o funcionamento dos nossos genes, e assim, abrir novos caminhos para a biologia, a medicina e a compreensão dos mecanismos básicos da vida.
Para estudar esses fenômenos, foram utilizadas técnicas de física computacional, que simplificam a enorme sequência de DNA em unidades menores, capazes de serem simuladas em computadores modernos com GPUs (placas gráficas de alto desempenho).
O estudo do artigo científico
No trabalho publicado na PNAS (10.1073/pnas.2530583123), os pesquisadores desenvolveram o Modelo de Cromatina de Inversão Completa (FI-Chrom), uma abordagem também guiada por dados experimentais. O FI-Chrom combina simulações aceleradas por GPUs com o princípio da máxima entropia (tendência de um sistema ir de um estado organizado para um desorganizado), mas com uma diferença fundamental: nesse caso, todo o conjunto de dados experimentais é utilizado de forma invertida, empregando também técnicas de minimização computacional e aprendizado de máquina.
O FI-Chrom é uma ferramenta computacional que cria modelos altamente precisos dos cromossomos, unindo física e inteligência artificial para compreender como o DNA se organiza no espaço tridimensional. Esse sistema permite conectar experimentos de laboratório a teorias físicas, possibilitando o estudo da dinâmica genética em diferentes organismos. Além disso, o modelo MiChroM permite simular o núcleo celular inteiro, podendo incluir as interações do DNA com outras estruturas internas e corpos nucleares, como nucléolos, speckles (manchas), e proteínas associadas à lâmina.
Entre os resultados, as simulações mostram que as alças de cromatina não são características arquitetônicas estáticas, mas sim elementos estruturais transitórios. Análises estatísticas indicam ainda que alças confinadas a um único compartimento ocorrem com mais frequência do que aquelas que abrangem múltiplos compartimentos, destacando a natureza dinâmica e dependente do compartimento da organização da cromatina.
O início da pesquisa
Em dezembro de 2022, o professor Ronaldo Oliveira foi para o Center for Theoretical Biological Physics (CTBP), referência mundial em biofísica teórica, na Rice University, em Houston, nos Estados Unidos, para realizar um pós-doutorado com duração de dois anos. Trabalhou com o professor José Onuchic, brasileiro, diretor do Centro e também coautor do artigo científico de doutorado, publicado na PNAS.

“Fui com apoio do CNPq, por meio de uma bolsa de pós-doutorado no exterior com duração de seis meses. No entanto, durante todo o período em que estive lá como pesquisador visitante, houve uma complementação financeira significativa como contrapartida da própria Rice University. Nesse período, me dediquei integralmente à pesquisa, tive a oportunidade de orientar alunos, colaborar em diferentes projetos e participar da organização e execução de workshops científicos”, relatou Ronaldo.
O objetivo inicial era aprender com o grupo do professor Onuchic as técnicas de simulação de cromatina, que representam os cromossomos que é uma mistura de DNA e proteínas localizada dentro do núcleo das nossas células. Para se ter uma ideia da escala, se o DNA de uma única célula fosse totalmente esticado, ele teria cerca de 2 metros de comprimento.
“Embora muitas ilustrações mostrem o cromossomo humano na forma de um X, com 23 pares, na maior parte do tempo o nosso DNA não está nessa forma. Ele se apresenta em um estado compactado chamado interfase, que é quando a célula não está em divisão. A grande pergunta científica, então, é: como os cromossomos se organizam dentro do núcleo celular? Essa é uma questão extremamente complexa. Imagine tentar mover 46 fios de linha extremamente finos, quilométricos, de um lado de uma sala para o outro, sem que nenhum se quebre ou se embaralhe. Isso seria praticamente impossível. Ainda assim, a compactação do DNA é tão eficiente que ele chega a ser reduzido em cerca de 10.000 vezes o seu tamanho original para caber dentro do núcleo das células. Essa arquitetura tridimensional dos cromossomos tem implicações diretas em processos fundamentais da vida, como a divisão celular, o controle da expressão gênica e a estabilidade genética. Ela também é crucial para o diagnóstico e compreensão de doenças e anomalias genéticas, como a Síndrome de Down e diferentes tipos de câncer”, explicou.
O primeiro trabalho, resultado dessas pesquisas foi um artigo publicado no Journal of Chemical Physics (JCP) (https://doi.org/10.1063/5.0239634), onde foram avaliados os potenciais e parâmetros de força do modelo de cromatina mínima (MiChroM), treinado com dados experimentais, com implicações diretas para a organização dos cromossomos na interfase. O MiChroM é parametrizado a partir de dados experimentais usando conceitos clássicos e bem estabelecidos da física estatística, como o princípio da máxima entropia, e descreve dois mecanismos fundamentais da organização nuclear: a compartimentalização de regiões do cromossomo e a motorização por proteínas organizacionais.
Foto: Arquivo pessoal de Ronaldo Oliveira/UFTM

Redes Sociais